Gaussian Mixture Model (GMM)
Gaussian Mixture Model (GMM)
From the Point of View of Geometry
对于观测到的样本,当然可以认为所有样本都属于同样一个高斯分布,但是这种观点有些时候是不合理的。从几何角度看,GMM认为这些样本属于多个高斯分布的加权平均:
\[p(x)=\sum_{k=1}^K\alpha_kN(x;\mu_k, \Sigma_k),\quad \sum_{k=1}^K\alpha_k=1\label{eq1}\]其中,$\alpha_k$是加权系数。
以一维随机变量为例:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
clc, clear, close all
rng("default")
% Specify parameters
x = -7:.1:7;
mu_1 = -2;
sigma_1 = 1;
mu_2 = 2;
sigma_2 = 2;
% The PDF of N1 and N2
y1 = normpdf(x, mu_1, sigma_1);
y2 = normpdf(x, mu_2, sigma_2);
% Sample points
x_1 = sigma_1*randn(50, 1) + mu_1;
x_2 = sigma_2*randn(50, 1) + mu_2;
% The PDF of Single Gaussian distribution
mu = mean([x_1; x_2]);
sigma = cov([x_1; x_2]);
Y = normpdf(x, mu, sigma);
% The weights of Gaussian Mixture PDF
alpha_1s = 0.1:0.2:0.7;
LineWidth = 1.5;
figure('Units', 'pixels', 'Position', [589 ,219, 1260, 1025], 'Color', 'w')
% Plot schematics
for idx = 1:4
nexttile
hold(gca, "on")
scatter(x_1, zeros(size(x_1)), 70, 'x', LineWidth=1)
scatter(x_2, zeros(size(x_2)), 70, '*', LineWidth=1)
plot(x, y1, LineWidth=LineWidth)
plot(x, y2, LineWidth=LineWidth)
alpha_1 = alpha_1s(idx);
alpha_2 = 1-alpha_1;
plot(x, alpha_1*y1+alpha_2*y2, 'k', LineWidth=LineWidth+0.5)
plot(x, Y, 'b', LineWidth=LineWidth+0.5)
legend('$x\in N_1$', '$x\in N_2$', '$N_1$', '$N_2$', 'Mixture N', 'Single N', Interpreter="latex")
xlabel('$x$', Interpreter="latex")
ylabel('PDF')
title(['Gaussian Mixture PDF, ', '$\alpha_1=$', num2str(alpha_1), ', $\alpha_2=$', num2str(alpha_2)], Interpreter="latex")
box on
grid on
end
exportgraphics(gcf, "fig.jpg", "Resolution", 900)
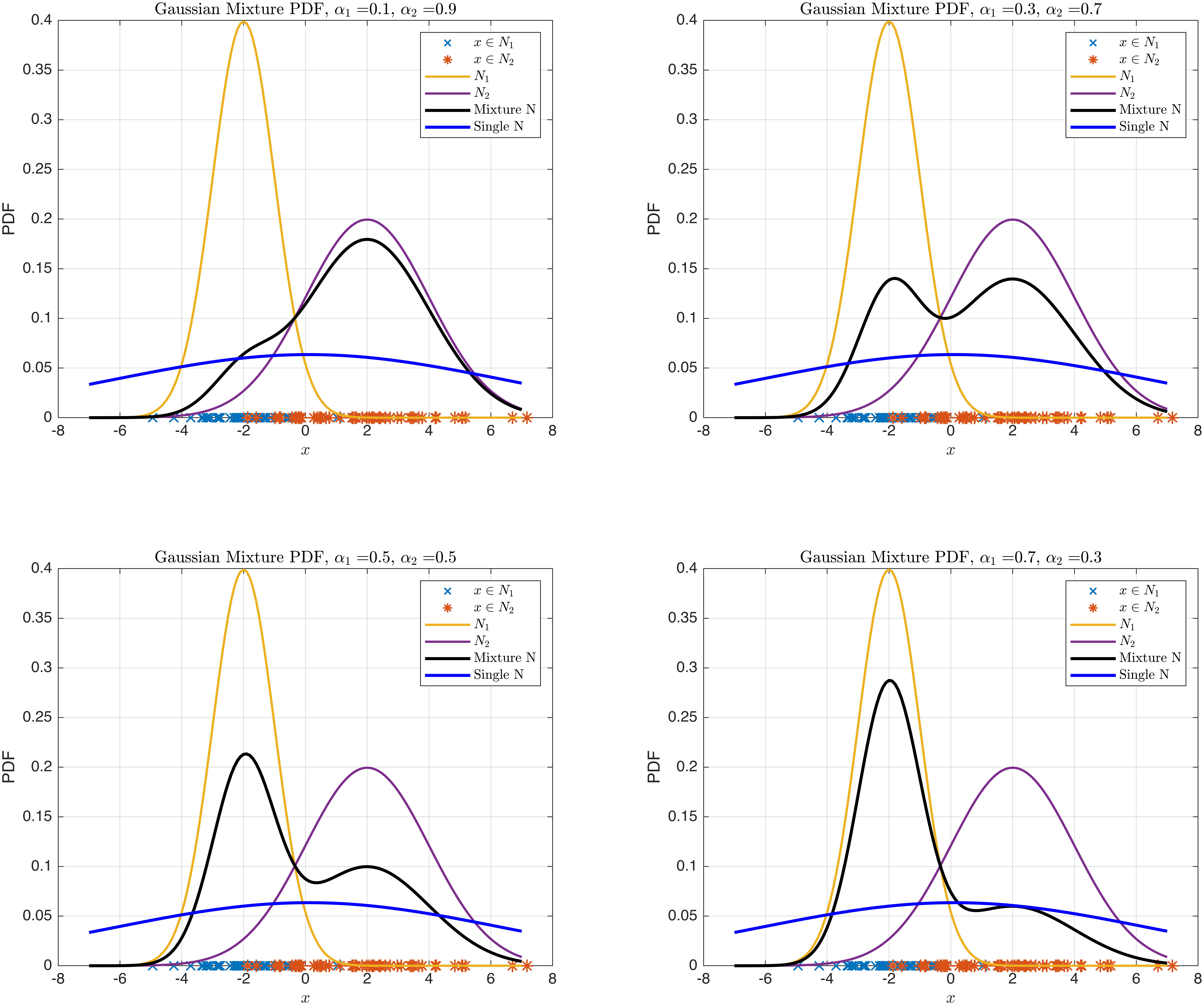
图中,黑线表示不同的高斯混合PDF,可以看到,选取不同的权重系数曲线是不同的;蓝线是认为样本属于单一的高斯分布而得到的PDF。
假设样本属于单一的高斯分布,那么很容易得到样本对应的PDF,只需要求出样本均值和样本协方差矩阵,带入到高斯分布的PDF中即可;而假设样本属于混合高斯分布,那么PDF是很多样的,并且在真实情况的情况下,每一个高斯分布的$\mu_k$和$\Sigma_k$都是未知的,权重系数$\alpha_k$也是未知的,必须通过复杂的过程确定最优的参数集合,这也是高斯分布模型的核心。
From the Point of View of Mixture Model
从混合模型角度来看,除了观测变量(observed variable)$x$之外,还需要引入一个隐变量(latent variable)$z$,隐变量$z$表示对应的样本$x$是属于哪一个高斯分布。隐变量$z$实际上一个离散随机变量,也属于一个分布,用于表征样本$x$属于某一高斯分布的概率。
| z | $c_1$ | $c_2$ | $\cdots$ | $c_k$ |
|---|---|---|---|---|
| p(z) | $p_1$ | $p_2$ | $\cdots$ | $p_k$ |
其中,$\sum_{k=1}^Kp_k=1$。
于是GMM所对应的PDF为(通过积分消去隐变量):
\[\begin{split} p(x)&=\sum_zp(x,z)\\ &=\sum_{k=1}^Kp(x,z=c_k)\\ &=\sum_{k=1}^Kp(z=c_k)p(x|z=c_k)\\ &=\sum_{k=1}^Kp_k\cdot p(x;\mu_k,\Sigma_k) \end{split}\label{eq2}\]式$\eqref{eq2}$和式$\eqref{eq1}$是一致的。
Estimation of Optimal Parameter Set of GMM
正如上文所述,GMM的核心就是根据(1)给定的样本和(2)给定的GMM component的个数,确定(1)每个GMM component的$\mu_k$和$\Sigma_K$,和(2)权重(或者说隐变量所对应的概率)的最优参数集合,这本质上是一个优化问题。
通过样本确定最优参数集合$\theta={p_1, \cdots,p_k,\mu_1,\cdots,\mu_k, \Sigma_1,\cdots,\Sigma_k}$,很自然地就想到使用极大似然估计MLE:
\[\begin{split} \hat{\theta}_{MLE}&=\arg\max\limits_{\theta}\log P(x)\\ &=\arg\max\limits_{\theta}\log\prod_i^Np(x_i)\\ &=\arg\max\limits_{\theta}\sum_i^N\log p(x_i)\\ &=\arg\max\limits_{\theta}\sum_i^N\log\Big[\sum_k^K p_k\cdot p(X_i;\mu_k,\Sigma_k)\Big] \end{split}\]但是,由于$\log$函数内部是多项连加,并且里面的MVN的PDF很复杂,这就导致GMM是没有解析解的,用极大似然估计是做不出来的。因此最优参数集合的求解需要用到数值算法,比如期望最大(Expectation-Maximization, EM)算法。
GMM in MATLAB
MATLAB官网提供了一个GMM拟合的示例,Cluster Using Gaussian Mixture Model1,该实例选用的数据集是Iris数据集,为了能够可视化,选取前两个特征:
1
2
3
4
5
6
7
8
9
10
11
clc, clear, close all
load fisheriris;
X = meas(:,1:2);
[n, p] = size(X);
figure(1)
plot(X(:,1), X(:, 2), '.', 'MarkerSize', 15);
title('Fisher''s Iris Data Set');
xlabel('Sepal length (cm)');
ylabel('Sepal width (cm)');

指定GMM component的个数和EM算法最大迭代数,以及协方差矩阵设置:
1
2
3
4
5
6
7
8
9
10
11
12
rng("default");
% Specify number of GMM components
k = 3;
% Specify maximum iterations for EM algorith
options = statset('MaxIter',1000);
% Specify coviarance structure options
Sigma = {'diagonal', 'full'}; % Options for covariance matrix type
nSigma = numel(Sigma);
SharedCovariance = {true, false}; % Indicator for identical or nonidentical covariance matrices
SCtext = {'true' ,'false'};
nSC = numel(SharedCovariance);
这些参数在后面都要传入到fitgmdist函数2中。
注:fitgmdist函数用的就是EM算法。
创建二维网格,供后续绘制每一个cluster的confidence ellipsoid图:
1
2
3
4
5
6
7
% Create a 2-D grid covering the plane composed of extremes of the measurements.
% Later use this grid later to draw confidence ellipsoids over the clusters.
d = 500; % Grid length
x1 = linspace(min(X(:,1))-2, max(X(:,1))+2, d);
x2 = linspace(min(X(:,2))-2, max(X(:,2))+2, d);
[x1grid, x2grid] = meshgrid(x1, x2);
X0 = [x1grid(:) x2grid(:)];
根据inverse cumulative distribution function (icdf) of the chi-square distribution指定阈值,后续会将网格点和该阈值进行比较,绘制不同的网格点:
1
2
3
% chi2inv, returns the inverse cumulative distribution function (icdf) of the chi-square distribution
% with degrees of freedom 2, evaluated at the probability values in 0.99.
threshold = sqrt(chi2inv(0.99, 2));
1
2
threshold =
3.0349
之后,创建循环遍历fitgmdist函数的每一种option,并进行绘图。每次循环主要进行的运算:
-
使用
fitgmdist函数拟合GMM,返回变量gmfit。1 2 3 4 5 6 7 8 9 10 11 12 13 14
gmfit = Gaussian mixture distribution with 3 components in 2 dimensions Component 1: Mixing proportion: 0.122116 Mean: 6.9393 3.0734 Component 2: Mixing proportion: 0.325422 Mean: 5.0153 3.4507 Component 3: Mixing proportion: 0.552462 Mean: 6.0888 2.8221
gmfit中包含着每个component的权重系数(即Mixing proportion),加和为1;以及每个component的均值信息。 -
利用
gmfit中的均值信息使用cluster函数对所有的数据点X进行聚类,并使用gscatter函数进行标记; -
计算网格点到每个component均值的马氏距离,并根据马氏距离和
threshold的关系进行网格点绘制。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
figure('Units', 'pixels', 'Position', [585, 307, 1120, 793], 'Color', 'w')
count = 1;
for i = 1:nSigma
for j = 1:nSC
gmfit = fitgmdist(X, k, 'CovarianceType', Sigma{i}, ...
'SharedCovariance', SharedCovariance{j}, 'Options', options); % Fitted GMM
clusterX = cluster(gmfit, X); % Cluster index
mahalDist = mahal(gmfit, X0); % Distance from each grid point to each GMM component
% Draw ellipsoids over each GMM component and show clustering result.
subplot(2, 2, count);
h1 = gscatter(X(:,1), X(:,2), clusterX);
hold on
for m = 1:k
idx = mahalDist(:, m)<=threshold;
Color = h1(m).Color*0.75 - 0.5*(h1(m).Color - 1);
h2 = plot(X0(idx, 1), X0(idx, 2), '.', 'Color', Color, 'MarkerSize', 1);
uistack(h2, 'bottom');
end
plot(gmfit.mu(:, 1), gmfit.mu(:,2), 'kx', 'LineWidth', 2, 'MarkerSize', 10)
title(sprintf('Sigma is %s\nSharedCovariance = %s', Sigma{i}, SCtext{j}), 'FontSize', 8)
legend(h1, {'1', '2', '3'})
hold off
count = count+1;
end
end
exportgraphics(gcf, "fig.jpg", "Resolution", 900)
最终得到不同fitgmdist函数option的结果和图像:
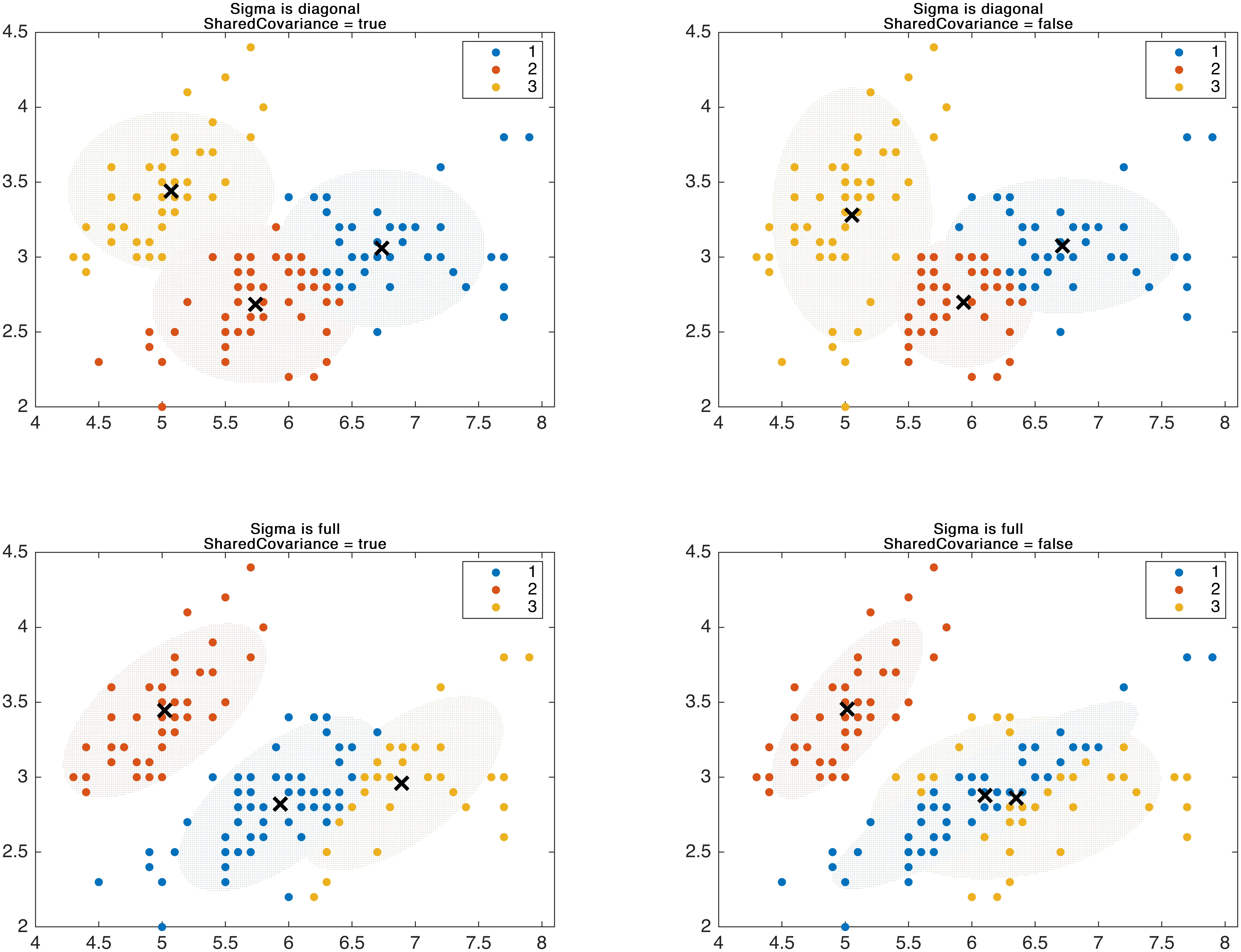
除此之外,fitgmdist函数还支持设置初始条件:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
% Record convergence situation
converged = nan(4, 1);
% For all instances, use k = 3 components, unshared and full covariance matrices,
% the same initial mixture proportions, and the same initial covariance matrices.
% For stability when you try different sets of initial values,
% increase the number of EM algorithm iterations. Also, draw confidence ellipsoids over the clusters.
for j = 1:4
gmfit = fitgmdist(X, k, 'CovarianceType', 'full', ...
'SharedCovariance', false, 'Start', cluster0{j}, ...
'Options', options);
clusterX = cluster(gmfit, X); % Cluster index
mahalDist = mahal(gmfit, X0); % Distance from each grid point to each GMM component
% Draw ellipsoids over each GMM component and show clustering result.
subplot(2, 2, j);
h1 = gscatter(X(:, 1), X(:, 2), clusterX); % Distance from each grid point to each GMM component
hold on;
nK = numel(unique(clusterX));
for m = 1:nK
idx = mahalDist(:,m)<=threshold;
Color = h1(m).Color*0.75 + -0.5*(h1(m).Color - 1);
h2 = plot(X0(idx, 1),X0(idx, 2), '.', 'Color', Color, 'MarkerSize', 1);
uistack(h2, 'bottom');
end
plot(gmfit.mu(:,1), gmfit.mu(:,2), 'kx', 'LineWidth', 2, 'MarkerSize', 10)
legend(h1, {'1', '2', '3'});
hold off
converged(j) = gmfit.Converged; % Indicator for convergence
end
sum(converged)
exportgraphics(gcf, "fig1.jpg", "Resolution", 900)

以上设置的GMM都是收敛的,但是各个设置所得到的结果还是有比较大的差异。另外,以上的GMM拟合都是非监督聚类,完全没有用到标签的信息,因此哪一种GMM都可以认为是合理的。但是,Iris是一个有标签的数据集,我们可以绘制一下真实的数据情况,用真实的情况作为参考:
1
2
3
4
5
clc, clear, close all
load fisheriris
X = meas(:, 1:2);
gscatter(X(:, 1), X(:, 2), species)

Generative Model
当我们用通过样本拟合出了一个GMM,这就意味着我们知道了GMM的PDF,于是就可以用这个PDF来生成样本数据了。从这个角度看,GMM也是一个生成模型,并且是一个很简单的生成模型,其简单程度仅次于单个高斯分布模型MVN-RNG(见博客3)。
GMM的概率图如下所示:

阴影部分为观测变量$x$,实心点表示参数。
References